ウイルスのゲノムやプラスミドのどの位置にPCRプライマーがくっつくのか(アニールするのか)、というプライマーマップを表示します。
検索対象となるプライマーは研究室内のプライマーDBにあるデータです(つまり、納品済みのもの)。
まずは、トップ画面。殺風景です。
プライマーの5’末端側に制限酵素を付加する場合もあるので3’末端から指定の塩基数だけ対象にすることもできます。イノシンや縮重塩基にも対応します。

とあるウイルスのゲノム配列をGenBankからコピペしてみます。数字や空白、改行がありますが問題ありません。

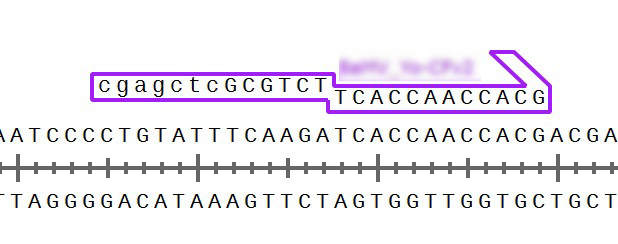
検索結果の一部。目的通りの位置にアニールすることが確認できました。
query配列(送信したゲノム配列)の全長を500ピクセルの線として上下に載せ、フォワードプライマーであれば→で、リバースプライマーであれば←で表現しています。プライマー名をクリックするとそのプライマーの詳細が表示されます。

ページの一番下に行くとSnapGeneViewer用にエクスポートできます。ファイル形式はGenBank形式なのでSnapGeneVewerに限らず、多くの解析ソフトで開けます。
ファイル名を記入し、線状か環状を選び、エクスポートをクリックするとファイルがDLされます。
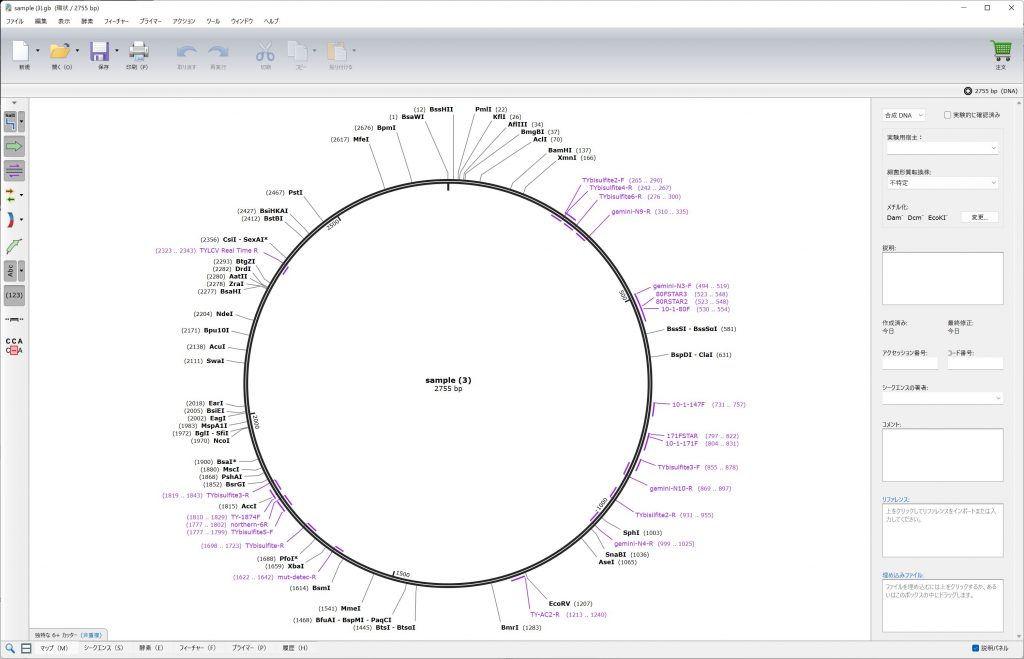
表示例。小さくて見づらいですが、赤紫色の文字がプライマー、黒い文字が自動で追加された制限酵素認識サイトです。
試しに3’末端から10塩基だけを使ってもう一度検索してみます。
当然ヒットするプライマーは増えます。
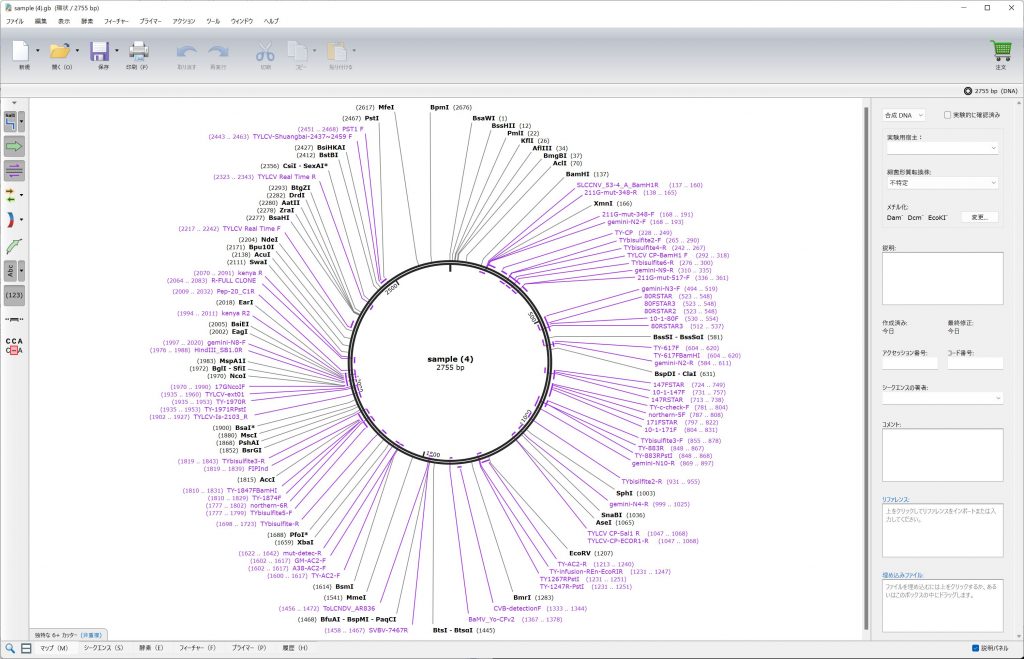
「シークエンス」で詳細を見ると、制限酵素認識サイトを付加したものもくっついていることが分かります。